数据库由BraTS、TCIA、湘雅医院脑肿瘤图像数据组成;现有299例病人的图像数据;肿瘤类型包括:
高级胶质瘤、低级胶质瘤、胶质母细胞瘤、
脑膜瘤等常见颅内肿瘤;BraTS数据总量达224,250张,其
中带标签数据共44,850张,TCIA数据库的数据包含的肿瘤类型较多,但是无标签数据,共1,568,986
张图片。
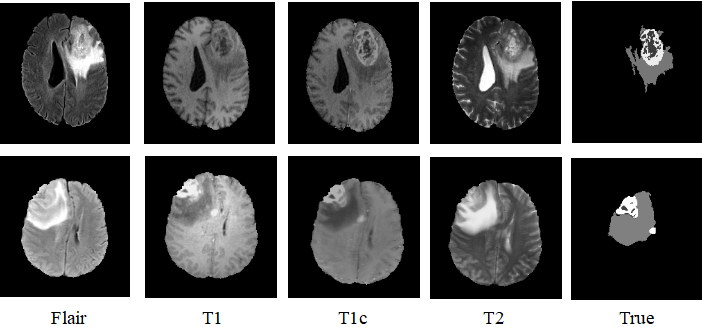
带标签的数据由四种模态数据,分别是Flair、T1、T1c、T2四种模态;不同模态下脑组织在图像中的
表现不同,T1模态下主要是看解剖结构,T2模态下观察病变区域,T1c观察脑组织中肿瘤的边缘。
BraTS数据库的四种模态的图像以及标签如下所示:
TCIA数据库的数据如下所示: